Die Regulierung von Zellprozessen beruht auf einem großen Pool an Transkriptionsfaktoren und ihren Wechselwirkungen mit DNA, also dem Erbmaterial. Dabei sitzen diese speziellen Proteine wie winzige Anker auf der DNA und ermöglichen die Abschrift genetischer Information. Trotz des weit verbreiteten medizinischen, pharmakologischen und biologischen Interesses sind viele wesentliche Merkmale der Wechselwirkungen zwischen den Transkriptionsfaktoren und dem Erbgut auch nach langjährigen Bemühungen noch immer wenig verstanden. Man spricht von dem "dunklen Proteom".
Dunkles Proteom
Der Wiener Methodenentwickler Dennis Kurzbach vom Institut für Biologische Chemie / NMR Core Facility konnte aufbauend auf integrative Magnetresonanz einen Ansatz finden, das Verhalten der ungeordneten Proteinabschnitte von Transkriptionsfaktoren zu beobachten.
Intrinsisch ungeordnete Proteine (IDPs = intrinsically disordered proteins) oder Proteinregionen zeichnen sich dadurch aus, dass ihnen charakteristische Strukturmerkmale fehlen. Im Gegensatz zu den Proteinen mit einer geordneten 3D-Struktur wie einer lang gesteckten Helix, die in der Interaktion mit ihren biologischen Partnern nach dem "Schlüssel-Schloss-Prinzip" funktionieren, sind die IDPs flexibler. Dass die evolutionäre Entwicklung von IDPs gute Gründe gehabt haben muss, dafür sprich schon allein ihr Anteil von bis zu 30 Prozent in der Gesamtheit der Proteine im menschlichen Organismus.
Strukturdynamik charakterisiert
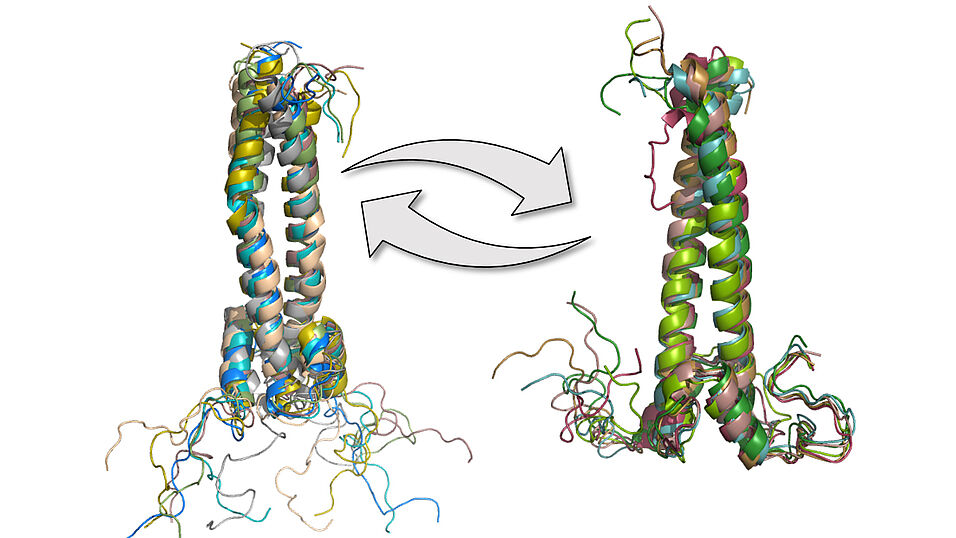
IDPs übernehmen wichtige Funktionen bei der Regulierung biologischer Prozesse. Sie sind z.B. wichtige Knotenpunkte im Kommunikationsnetzwerk zwischen DNA und Proteinen, einem sehr dynamischen System. In der vorliegenden Studie konnte nun das Team um Dennis Kurzbach die Strukturdynamik jener intrinsisch gestörten Region charakterisieren, die die DNA-Bindungsstelle von dem lebenswichtigen Transkriptionsfaktors MAX (engl. Myc-Associated factor X) ist.
"Unsere Beobachtungen waren durch eine Integration von Kernspinresonanz-Messungen, Molekulardynamik-Simulationen und Messungen der Elektronenspinresonanz möglich – das ist das Wunderbare: Die heutigen Technologien ermöglichen es uns, als Toolmaker ganz neue Methoden und Ansätze zu entwickeln, um lange Zeit unüberwindbare Barrieren zu überwinden und strukturbiologischen Fragestellungen in ganz neuen Dimensionen von Raum und Zeit nachzugehen", so Dennis Kurzbach:
"Das daraus gewonnene Verständnis hilft uns u.a. dabei, grundlegende Prozesse in Zellen oder bei der Tumorgenese zu verstehen – und später vielleicht einmal auch neue Ansätze beim Medikamentendesign und Behandlungsstrategien zu finden."
S E R V I C E:
A Switch between Two Intrinsically Disordered ConformationalEnsembles Modulates the Active Site of a Basic-Helix−Loop−HelixTranscription Factor. Giuseppe Sicoli, Thomas Kress, Hervé Vezin, Karin Ledolter, and Dennis Kurzbach, J.Phys.Chem.Lett.2020, 11, 8944−8951, published on 8 October 2020, https://doi.org/10.1021/acs.jpclett.0c02242